Général
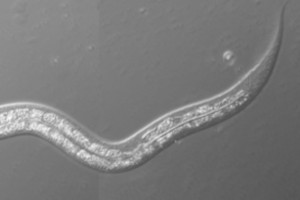
Les avantages de l’utilisation de Caenorhabditis elegans (C. elegans) en tant qu’organisme modèle sont divers et variés. Tout d’abord, le développement respecte un programme invariant d’un individu à l’autre, et est parfaitement connu. Cette connaissance est particulièrement utile pour étudier les mécanismes de différenciation cellulaire.
De la même façon, un nombre précis de cellules meurent au cours du développement, ce qui facilite l’étude des mécanismes intervenant dans l’apoptose. La nature endogamique de C. elegans par auto-fécondation et la possibilité de croiser les hermaphrodites avec les mâles offrent un avantage jusque là réservé au système de reproduction des plantes. Les souches sont de plus faciles à élever et conserver à long terme.
C. elegans est aussi un des plus simples organismes comportant un système nerveux, permettant une étude aisée de ce système. Enfin, son corps transparent facilite l’étude du développement et de la différentiation cellulaire qui est proche de celle des vertébrés.
Reproduction
Période de reproduction: toute l’année.
Cycle de vie : à 22°C, env. 55 heures entre l’œuf et l’adulte.
Deux sexes : mâles et hermaphrodites.
Les hermaphrodites peuvent se reproduire avec les mâles ou s’auto-féconder.
Outils
- Banque d’ESTs qui a permis d’améliorer l’annotation du génome et d’estimer l’efficacité des prédictions de gènes
- L’ORFéome (totalité des Open Reading Frames (ORF) du génome prédits in silico) dont les données sont regroupées dans la base de données Worfdb
- Le Promotérome : banque de clones contenant les promoteurs prédits in silico dans des vecteurs flexibles de type Gateway™ (Invitrogen), ce qui permet l’intégration de ces promoteurs dans d’autres vecteurs. Les clones sont disponibles dans une base de données spécifique
- La mise en place de l’ORFéome et du Promotérome a rendu possible la mise en place d’un grand nombre de techniques et d’analyse de génomique fonctionnelle pour C. elegans (transcriptome, localisation exacte de protéines…)
- L’extinction de gènes par RNAi permet l’analyse à grande échelle de la fonction des gènes chez C. elegans. Tous les phénotypes RNAi sont à la disposition des chercheurs sur la banque public Wormbase
- Le C. elegans Knockout Consortium conduit à des analyses d’extinction de gènes en réalisant la délétion d’allèles de certains gènes cibles
- Des profils d’expression ont été obtenus grâce à des organismes génétiquement modifiés avec des constructions promoteur::GFP ou promoteur::ORF::GFP
- Le Génome Science Center met à disposition des chercheurs une banque de profils d’expression obtenus à partir de la construction promoteur::GFP
- La cartographie des interactions protéine-protéine a été établie grâce au système de « double hybride », dans le cadre de projet « Interactome Mapping Project ». Les données sont regroupées dans la base de données Worm Interaction Sequences Tags database
- Plusieurs études ont analysé l’expression génique par RNAseq avec une résolution de cellule unique (par ex. http://atlas.gs.washington.edu/worm-rna/)
Bases de données
Infrastructures
- Plateforme de Génomique Fonctionnelle de C. elegans - Maïté Carre-Pierrat (Lyon) et Jonathan Ewbank (Marseille)
- http://www.ciml.univ-mrs.fr/technology/c-elegans-functional-genomics
- https://www.ibisa.net/plateformes/detail.php?tri=&srch=elegans&q=295
- Lyon et Marseille
- Liste des laboratoires travaillant sur le nématode en France
- http://www.ciml.univ-mrs.fr/EWBANK_jonathan/vermidi/labs.html
- https://wormbase.org/resources/laboratory#2--10
Experts
- Jonathan EWBANK
- ewbank@ciml.univ-mrs.fr
- CNIL, Marseille