Général
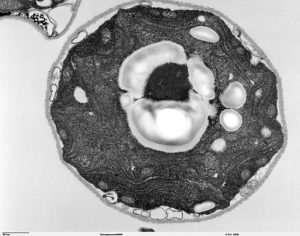
Chlamydomonas reinhardtii est une microalgue verte unicellulaire de 6-8 μm appartenant à la division des Chlorophytes et à l’ordre des Volvocales qui se trouve dans un large gamme d’environnements y compris les étangs, les marais, les volcans, les montagnes et la mer. Les caractéristiques essentielles du genre sont deux flagelles antérieurs de longueur égale qui émergent du corps cellulaire, une paroi cellulaire, un stigma ou « tache-oculaire » qui sent la lumière et un seul chloroplaste contenant un ou plusieurs pyrénoïdes, le site du mécanisme de concentration du CO2. Le chloroplaste simple de Chlamydomonas occupe jusqu’à 75 % du volume cellulaire et contient plusieurs copies (~ 80) d’ADN circulaire d’une taille d’environ 200 kb codant pour un total d’environ 100 protéines.
Chlamydomonas est utilisée comme système modèle pour la recherche biomédicale sur le développement et fonctionnement des flagelles. C. reinhardtii a été également largement utilisé comme organisme modèle pour la recherche sur la photosynthèse parce qu’il s’agit d’un photoautotrophe facultatif. Les mutants qui sont affectés dans le processus de photosynthèse peut se développer de manière hétérotrophe sur une source de carbone, l’acétate, et peuvent développer verte à l’obscurité parce qu’il existe une voie de biosynthèse de la chlorophylle qui est indépendante de la lumière chez cette espèce. Plus récemment, ce modèle a été utilisé comme modèle pour les études sur les biocarburants de troisième génération et le mécanisme de concentration du carbone.
- Nom : Chlamydomonas reinhardtii
- Classification phylogénétique : algue verte unicellulaire de la classe des Chlorophycées, de la famille des Chlamydomonadaceae
- Génome séquencé (2007)
-
Taille du génome : 120 millions de paires de bases.
Nombre de gènes prédits : environ 15.000 genes.
Reproduction
Chlamydomonas présente un cycle de vie haplobiontique en ce sens qu’il se reproduit de manière asexuée par division mitotique des cellules haploïdes. Le cycle cellulaire peut être synchronisé en alternant des périodes de lumière et d’obscurité, mais souvent en laboratoire les cellules sont maintenues sous une lumière continue qui rend leur division asynchrones.
Sous l’effet d’une carence d’azote, des gamètes haploïdes se développent. Il existe deux types de cellules reproductrices d’apparence identique plus (mt+, maternelle) et moins (mt-, paternel), qui peuvent fusionner pour former un zygote diploïde méiotique dans la plupart des cas (diploïde végétatif ou les cytoductants haploïdes sont présents dans <10% des zygotes). Le zygote n’est pas flagellé, et il représente une forme dormante des cellules. À la lumière, le zygote subit une méiose et libère quatre des cellules haploïdes flagellées qui reprennent le cycle de vie végétative. En laboratoire, l’ensemble du cycle de reproduction peut être réalisé en l’espace d’un mois.
Outils
- Mutagenese classique par des méthodes UV ou EMS
- Transformation nucléaire par l'insertion aléatoire d'un transgène par électroporation ou agitation avec des billes de verre
- RNAi
- Transformation de génomes chloroplastique ou mitochondrial par bombardement.
- TILLING
- CRISPR-Cas9
- Collection de marqueurs génétiques
- Collections de mutants
- Librairie des BACs, Cosmids et Fosmids
- MoClo
Bases de données
Plants.ensembl.org : https://plants.ensembl.org/Chlamydomonas_reinhardtii/Info/Index
Phytozome : https://phytozome.jgi.doe.gov/pz/portal.html#!info?alias=Org_Creinhardtii
Proteome : https://www.uniprot.org/proteomes/UP000006906
Adressages : https://giavap-genomes.ibpc.fr/
Collection des mutants : https://www.chlamycollection.org/
Infrastructures
- Institut de Biosciences et biotechnologies d’Aix-Marseille (BIAM) – CEA Cadarache
- http://biam.cea.fr/drf/biam/Pages/accueil.aspx
- Saint-Paul-lez-Durance
- Biologie du Chloroplaste et Perception de la Lumière chez les Micro-algues (UMR7141) – IBPC
- http://www.ibpc.fr/UMR7141/en/home/
- Paris
- Laboratoire Physiologie Cellulaire et Végétale (LPCV) – CEA Grenoble
- http://www.lpcv.fr/
- Grenoble
- Bioénergétique et Ingénierie de Protéines (BIP UMR7281) – Marseille
- https://bip.cnrs.fr/
- Marseille
- Synthetic and Systems Biology of Microalgae, Biologie Computationnelle et Quantitative UMR 7238 CNRS - Sorbonne Université
- http://www.lcqb.upmc.fr/content/synthetic-and-systems-biology-microalgae
- Paris
Experts
- Xenie JOHNSON
- xenie.johnson@cea.fr
- UMR7265 CEA-CNRS-AMU DRF / Cadarache CEA Research Center F-13108 Saint-Paul-lez-Durance, France
Bibliographie
- Livres
The Chlamydmonas Source Book. 2nd Edition, 2009.
Edited by Elizabeth H. Harris, David B. Stern, George B. Whitman.
https://www.sciencedirect.com/book/9780123708731/the-chlamydomonas-sourcebook
- Articles
The Chlamydomonas genome reveals the evolution of key animal and plant functions.
Merchant SS, Prochnik SE, Vallon O, Harris EH, Karpowicz SJ, Witman GB, Terry A, Salamov A, Fritz-Laylin LK, Marchal-Drouard L et al. 2007. Science. 318:245-250.
Birth of a Photosynthetic Chassis: A MoClo Toolkit Enabling Synthetic Biology in the Microalga Chlamydomonas reinhardtii.
Crozet P, Navarro FJ, Willmund F, et al. ACS Synth Biol. 2018;7(9):2074-2086.
An Indexed, Mapped Mutant Library Enables Reverse Genetics Studies of Biological Processes in Chlamydomonas reinhardtii.
Li X, Zhang R, Patena W, et al. Plant Cell. 2016;28(2):367-387.
Historical perspective on Chlamydomonas as a model for basic research: 1950-1970.
Goodenough U. Plant J. 2015;82(3):365-369.
- Ressources web
International Conference on the Cell and Molecular Biology of Chlamydomonas (Chlamy) : https://chlamy2020.sciencesconf.org/
Page Wikipédia : https://fr.wikipedia.org/wiki/Chlamydomonas_reinhardtii